使用Geneious快速定位基因魔剪CRISPR/cas的gRNA位點與脫靶結(jié)合位點[2]
基于機(jī)器學(xué)習(xí)翻譯,僅供參考
練習(xí)1:查找CRISPR位點
在本練習(xí)中,您將運行Find CRISPR位點功能來查找LYP1 CDS序列上的“GN(20)GG”CRISPR網(wǎng)站。
選擇LYP1 CDS(這個文件是geneious專用的demo數(shù)據(jù)格式,當(dāng)您下載了geneious可以導(dǎo)入進(jìn)去使用,或者導(dǎo)入您感興趣領(lǐng)域的數(shù)據(jù))文檔,然后轉(zhuǎn)到克隆→查找CRISPR位點。
首先,將選項重置為默認(rèn)設(shè)置。要做到這一點,點擊窗口左下角的齒輪,并選擇重置為默認(rèn)值(如果當(dāng)前設(shè)置為默認(rèn)設(shè)置,這將變?yōu)榛疑?/font>
由于我們想要在此序列中的任意位置查找CRISPR位點,因此在綁定位點旁依次選擇無處不在。
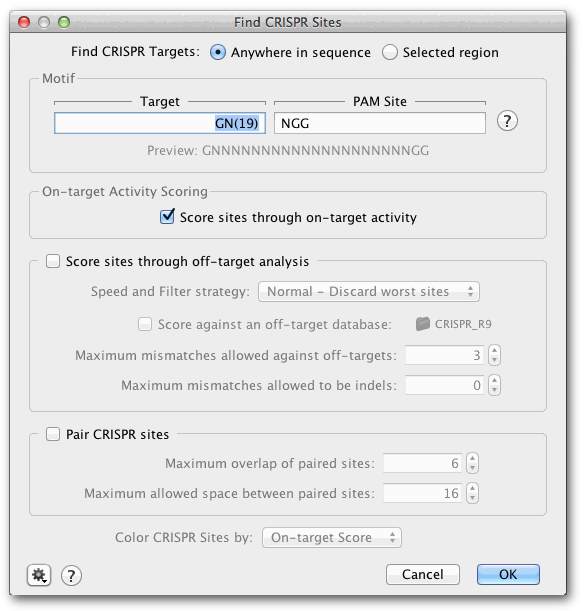
在Motif面板中,使用Target和PAM字段指定要搜索的gRNA序列。如果您希望評估所有潛在的CRISPR位點,請在目標(biāo)框中輸入N(20)。或者,如果您想要檢查異地綁定的特定序列,則可以在目標(biāo)框中輸入確切的序列。在本教程中,我們對“GN(20)GG”指南序列感興趣。目標(biāo)序列為GN(19),PAM序列為NGG,因此目標(biāo)字段類型為“GN(19)”,PAM Site字段類型為“NGG”。這些字段下方的預(yù)覽顯示了該目標(biāo)和PAM序列的指導(dǎo)序列 - 即GNNNNNNNNNNNNNNNNNNNNGG
在本練習(xí)中,我們只關(guān)注“GN(20)GG”位點在序列中的位置,因此取消選中通過脫靶分析對位點進(jìn)行評分的選項。我們要評估每個位點的活動,因此請通過檢查目標(biāo)活動來保持分?jǐn)?shù)網(wǎng)站。對話框現(xiàn)在應(yīng)該如下圖所示。點擊確定運行搜索。
 ?
?
Geneious將使用“GN(20)GG”基序鑒定該序列上的任何CRISPR位點并對其進(jìn)行注釋。“查找CRISPR位點”功能每次運行的注釋都被分組到一個標(biāo)有CRISPR位點的新軌道中。CDS中有41個“GN(20)GG”CRISPR位點。
將鼠標(biāo)懸停在其中一個網(wǎng)站上,您將看到一個彈出式窗口,其中包含有關(guān)該網(wǎng)站的信息。該信息包括On-target活動評分,其是在該位點的gRNA結(jié)合和Cas9切割效率的量度,如在Doench等2014中所計算的。此分?jǐn)?shù)是介于0和1之間的數(shù)字,數(shù)字越高表示預(yù)期活動越高。CRISPR位點注釋根據(jù)此分?jǐn)?shù)自動著色,從綠色(高分)到黃色和紅色(差分)的漸變。
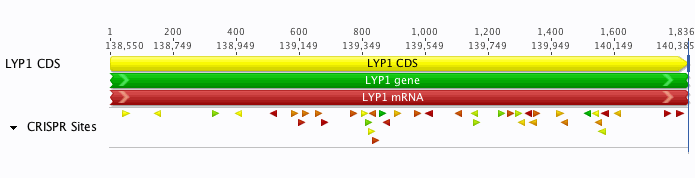

要從序列中刪除這些注釋,請單擊序列視圖中曲目名稱左側(cè)的箭頭,然后選擇刪除曲目。
未完,繼續(xù)第三[3]部分